
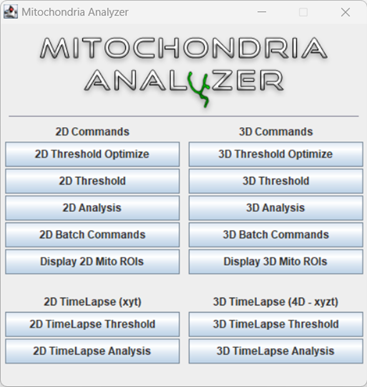
线粒体是具有复杂结构的细胞器,在研究线粒体时经常需要对线粒体的形态、结构进行分析,我们在这里分享一个可以执行线粒体结构分析的Image/Fiji插件,Mitochondria Analyzer可支持复杂的线粒体网络的2D,2D时间序列图像分析,3D以及3D时间序列图像分析
1、 Mitochondria Analyzer插件安装方法
下载链接:https://github.com/AhsenChaudhry/Mitochondria-Analyzer
安装步骤:下载并打开 MitochondriaAnalyzer.zip 文件,并将其内容复制到 ImageJ/FIJI“plugins”文件夹中。这包括 MitochondriaAnalyzer 文件夹以及上面列出的依赖插件。
然后下载 opencv-windows-x86_64.jar 文件并粘贴到 plugins-> 的 Adaptive Thresholding 文件夹中。
注意:对于 MacOS 用户,请从以下位置安装更新的 adaptiveThr_.class 和 opencv-macosx-x86_64.jar 文件:https://github.com/qztseng/imagej_plugins/tree/master/current
imagescience.jar文件(3D ImageJ Suite 所需)必须单独下载
2、 图像处理
(1) 如果图像为多通道,需要拆分单通道,通过Image-Color-Split Channels拆分,要设置图像的pixel size,并将像素单位改成μm,通过Analyze->Set Scale设置,此外还要将图像转化为8-bit格式。
注:图像格式需要为tif格式,不可以是PNG或者Jpg图像,程序会报错。
(2) 打开Mitochondria Analyzer插件
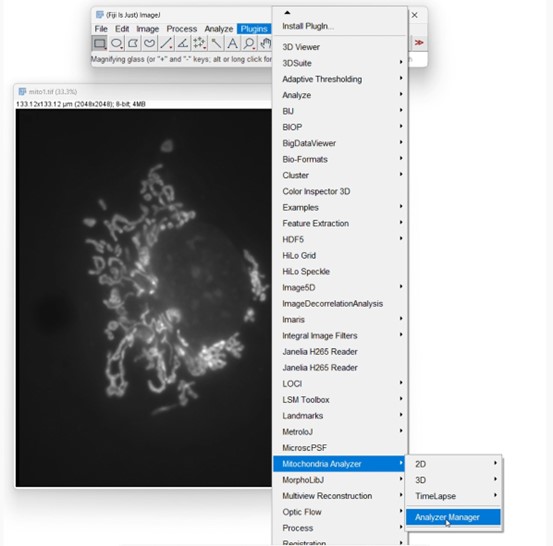
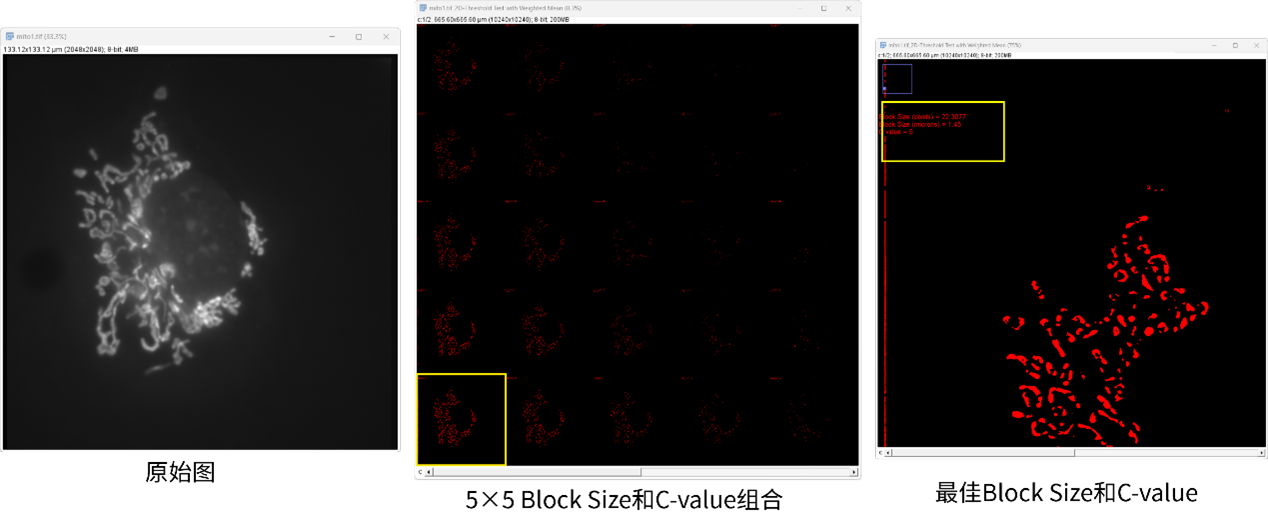
(3) 点击进入2D Threshold Optimize,可以看到阈值设定和图像处理过程,这里的参数是默认参数,根据算出的结果来计算最佳阈值组合,即Block Size和C-value
(4) 确定最佳参数组合后,点击进入2D Threshold或3D Threshold,对图像进行阈值化处理,以获得二值化图像,同时勾选Show comparison of threshold to original和Automatically perform analysis,计算完二值化图像后可直接得到图像的分析结果。
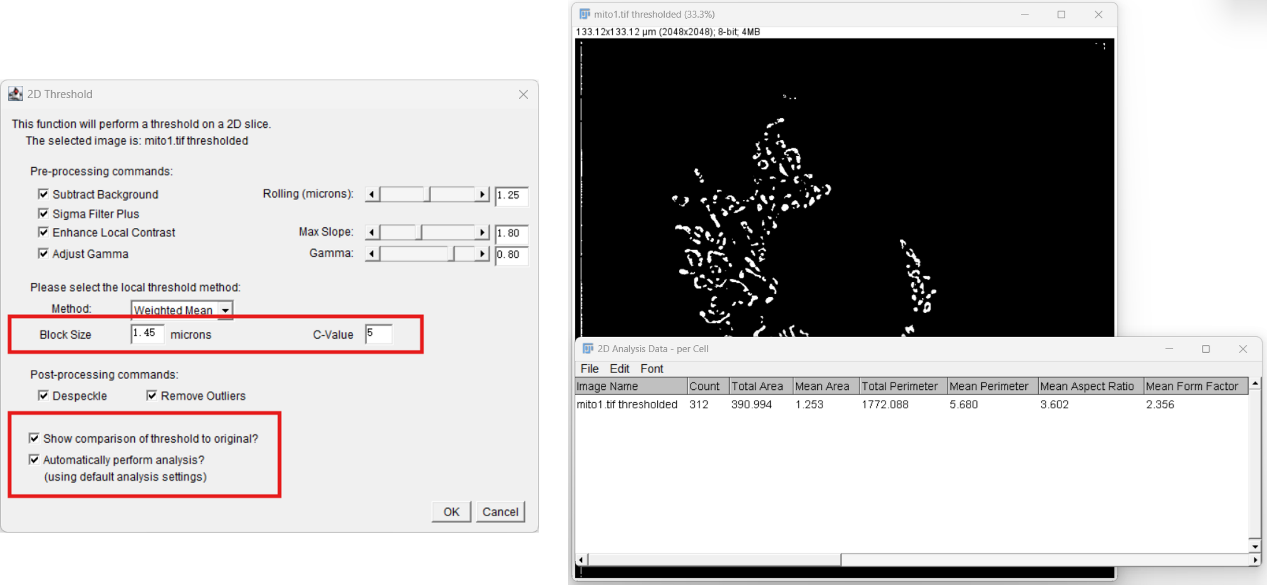
对于2D图像参数结果包括用以描述线粒体大小的线粒体面积(area)和周长(perimeter),以及用以描述形态的外形因数(form factor)和纵横比(aspect ratio)。描述线粒体网络的参数包括网络分支数(branches)、分支长度(branch lengths)、分支连接(branch junctions)。
(5) 或者Display 2D Mito ROIs将每一条线粒体分开,可以单个线粒体点击measure,也可以将所有的线粒体measure后可以得到每一条线粒体的信息


